识别组织切片是癌症或者正常。
数据:https://www.kaggle.com/c/histopathologic-cancer-detection/data
组织切片数据,来识别是癌症组织还是正常组织,我觉得是没多大意义的。因为在临床上,基本不可能获得一个病人的组织切片来检测是否患病,检测是否患病的代价太大是没有意义的。组织切片更多的是研究机体组织层面的情况,来研究细胞的作用机制,形态等。
这里就不深究。
网站给的数据非常庞大,训练集20多w张图片,测试集也有6w多。由于我的电脑太菜,我只用了1w张作训练集。
给的图片非常规整,全部为96 * 96像素的。因为想直接借鉴lenet5结构,我试了下直接resize成32 * 32的图片,但我自己感官上看图片变的非常模糊,96 / 32 =3, 损失了2/3的信息,我觉得可能效果不理想,就没有这样做。直接保存了96 * 96像素的大小。
先来看一下数据:
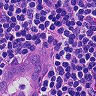
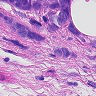
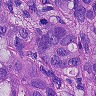
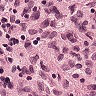
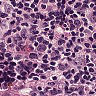
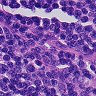
虽然有细胞生物学和生物化学以及分子生物学的基础,但是我没有真正看过组织切片,一开始也是没有看出正常和癌症的区别。后来由癌细胞的特性,观察可能是癌症组织会入侵到正常组织中,造成细胞形态不正常,还有大小吧。。。不是很清楚。染色什么的也不能分辨是癌症还是正常组织。总之,对一个没有看切片经验的人来说,还是比较困难的。
这次分类用的结构和之前的猫狗大战一样,是改良版的vgg16。不知道的小伙伴可以看往期的专栏,那里有改良版的vgg16的结构。
但是由于输入像素为96*96*3的图片,比猫狗大战的64*64*3多的很多,导致训练比较慢,总共训练了不到1w轮,测试集85%左右的正确率。
做了3个tensorflow的实战,还是有一些感触的。
总体来说,卷积步骤能够赋予大多数网络一定的学习的能力,但是,个人的网络往往学习能力都不强。这时候不得不借助成熟的经典网络,但是,随之而来的问题是经典网络往往对输入图片的大小都有要求,在转换的过程中,会损失或者失去很多信息。比如说原vgg16的输入是224 * 224 * 3,如果将组织切片数据转换为224,则会加入一些预测的信息,这一定会对结果造成影响。由大图片转换为小图片更可能丢失信息。再者,成熟的网络大多数都是深度网络,卷积池化和全连接加起来有接近20层,这在pc上很难调试和训练(没试过GPU)。
最近看了一篇迁移学习的文章,很有用,可能会试试看。




Twitter
Facebook
Reddit
LinkedIn
StumbleUpon
Pinterest
Email